文章已经过时,请去官网查阅相关文档
简介
在发育过程中,细胞对刺激作出反应,并在整个生命过程中,从一种功能“状态”过渡到另一种功能“状态”。不同状态的细胞表达不同的基因,产生蛋白质和代谢物的动态重复序列,从而完成它们的工作。当细胞在状态之间移动时,它们经历一个转录重组的过程,一些基因被沉默,另一些基因被激活。这些瞬时状态通常很难描述,因为在更稳定的端点状态之间纯化细胞可能是困难的或不可能的。单细胞RNA-Seq可以使您在不需要纯化的情况下看到这些状态。然而,要做到这一点,我们必须确定每个cell在可能的状态范围内的位置。
Monocle介绍了利用RNA-Seq进行单细胞轨迹分析的策略。Monocle不是通过实验将细胞纯化成离散状态,而是使用一种算法来学习每个细胞必须经历的基因表达变化序列,作为动态生物学过程的一部分。一旦它了解了基因表达变化的整体“轨迹”,Monocle就可以将每个细胞置于轨迹中的适当位置。然后,您可以使用Monocle的微分分析工具包来查找在轨迹过程中受到调控的基因,如查找作为伪时间函数变化的基因一节所述。如果这个过程有多个结果,Monocle将重建一个“分支”轨迹。这些分支与细胞的“决策”相对应,Monocle提供了强大的工具来识别受它们影响的基因,并参与这些基因的形成。在分析单细胞轨迹中的分支的小节中,您可以看到如何分析分支。
monocle 能做的不只是拟时分析,或者说为了做拟时分析他也做了sc-rna-seq的基本分析流程:数据读入,均一化,降维(PCA,umap,tsne,),聚类,marker基因筛选以及可视化函数。在新的学习中我们发现monocle能做的远不只这些,例如用shiny开发了web程序,更加用户友好;借助garnett包可以做细胞定义—–monocle已经是一个sc-rna-seq数据分析的工具箱。
Monocel对象的生成
Monocle的cds对象其实在一定程度上非常类似于Seurat,只不过表示方法不一样,所以,我们可以很容易的从Seurat中取出需要的数据载入Monocle3
具体方法可以参考简书:scRNA-seq数据分析 || Monocle3
但是,monocle软件有自己的一套流程,囊括了标准化,归一化,降维,聚类等等,所以一般来说,我们都需要提供原始的未经处理的表达矩阵,但是,由于我们是整合了多样本的结果,而我们又不想使用monocle的批次效应去除方法,我们该怎么导入呢?
方法就是我们导入Seurat多样本整合并标准化的结果矩阵,然后,在后面的预处理过程中,取消掉标准化。
1 | library(Seurat) |
预处理
数据scale并采用PCA降纬
1 | #we use normalized data,so we do not normalize it |
如果你非要使用Monocle的批次效应功能,可以尝试,batch列必须在你的pData里面
1 | cds <- align_cds(cds, alignment_group = "batch") |
采用umap的方法运行非线性降维
1 | #umap |
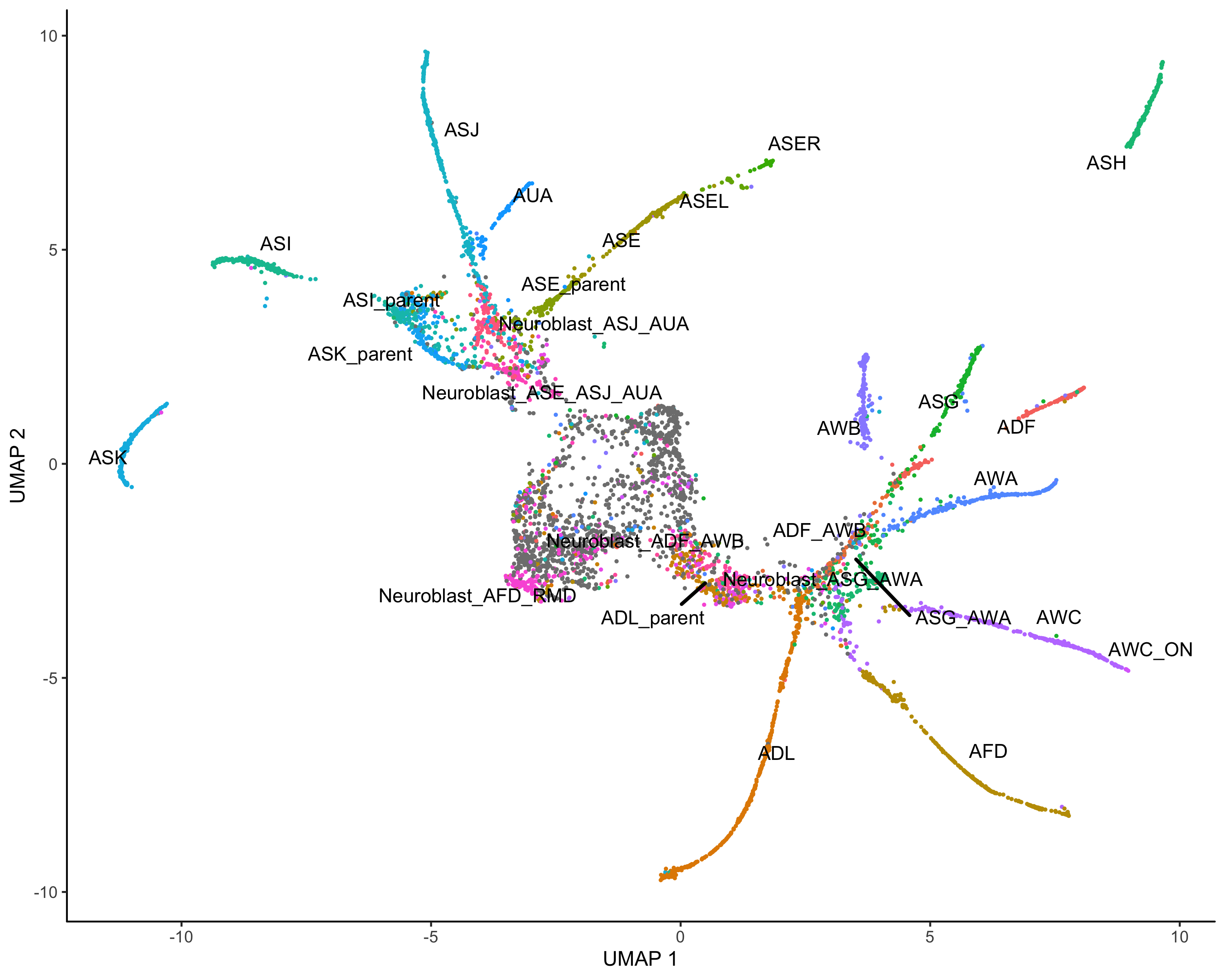
细胞聚类
1 | #cluster |
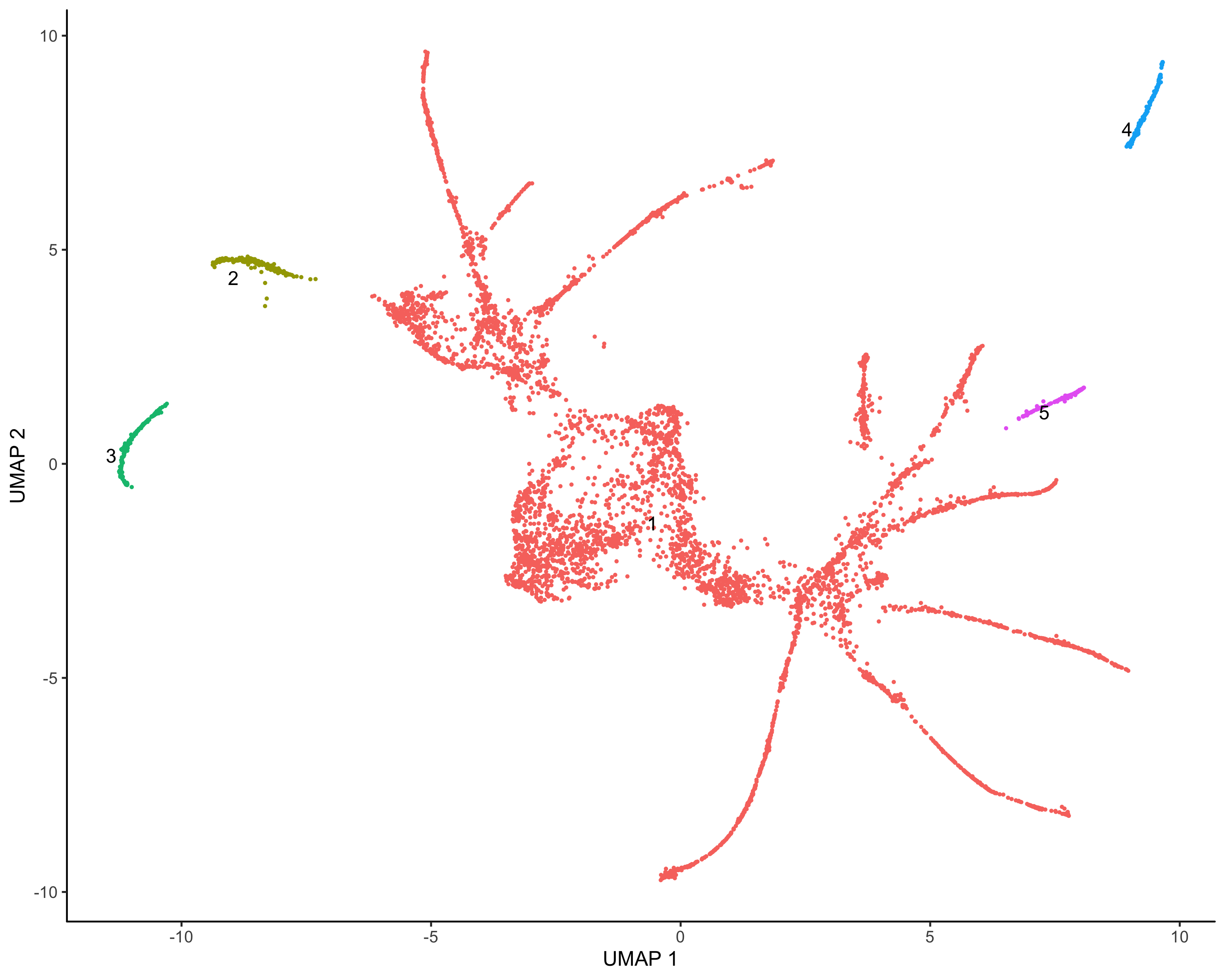
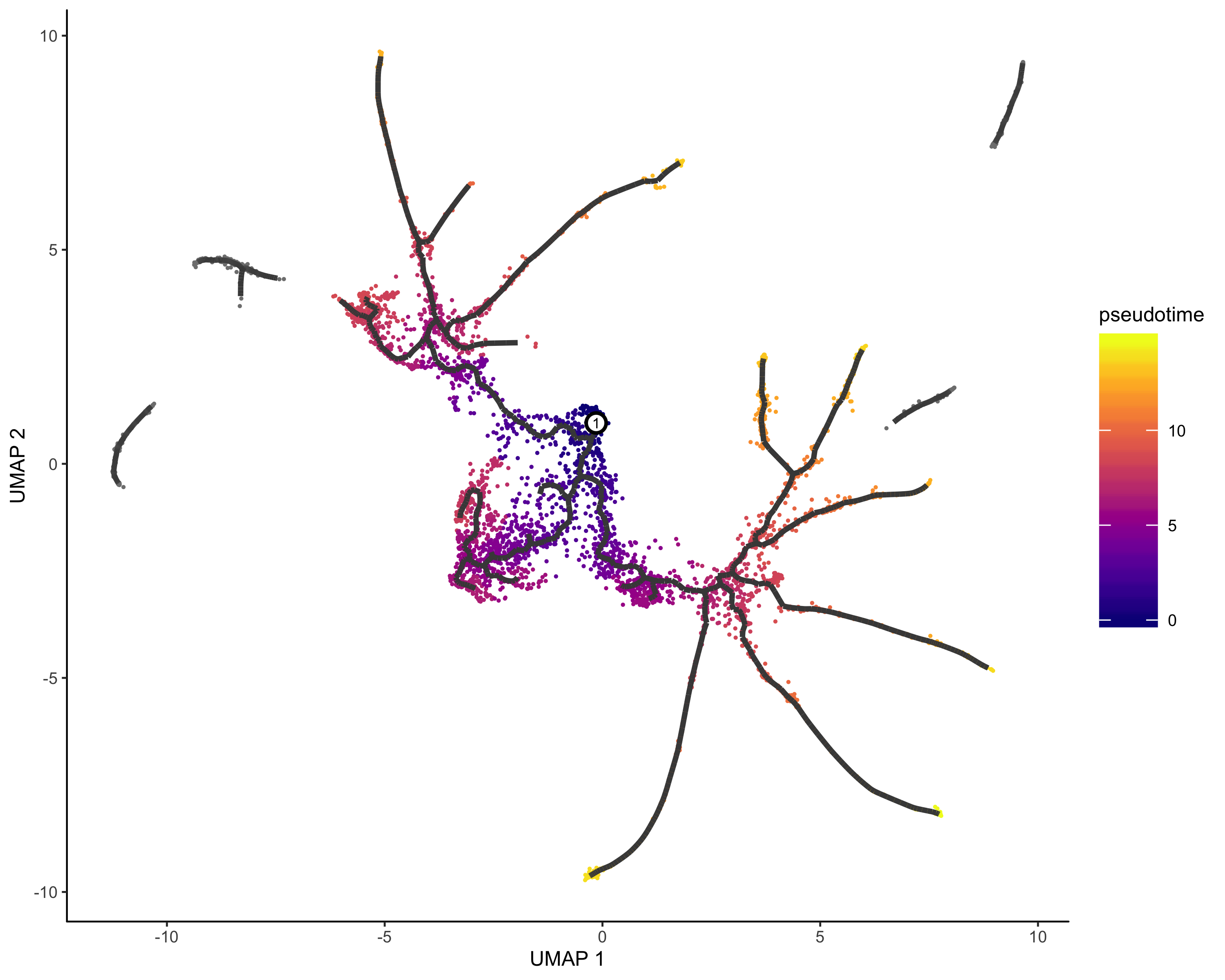
伪时间构建
其实说白了,就是基于图形以及表达量的变化关系,构建一个个基于起始表达的进化线或者说是分支,这里面肯定既有节点又有分枝。
1 | cds <- learn_graph(cds) |
画图展示:
1 | plot_cells(cds,color_cells_by = "Cell.type",label_groups_by_cluster=FALSE,label_leaves=TRUE,label_branch_points=TRUE) |
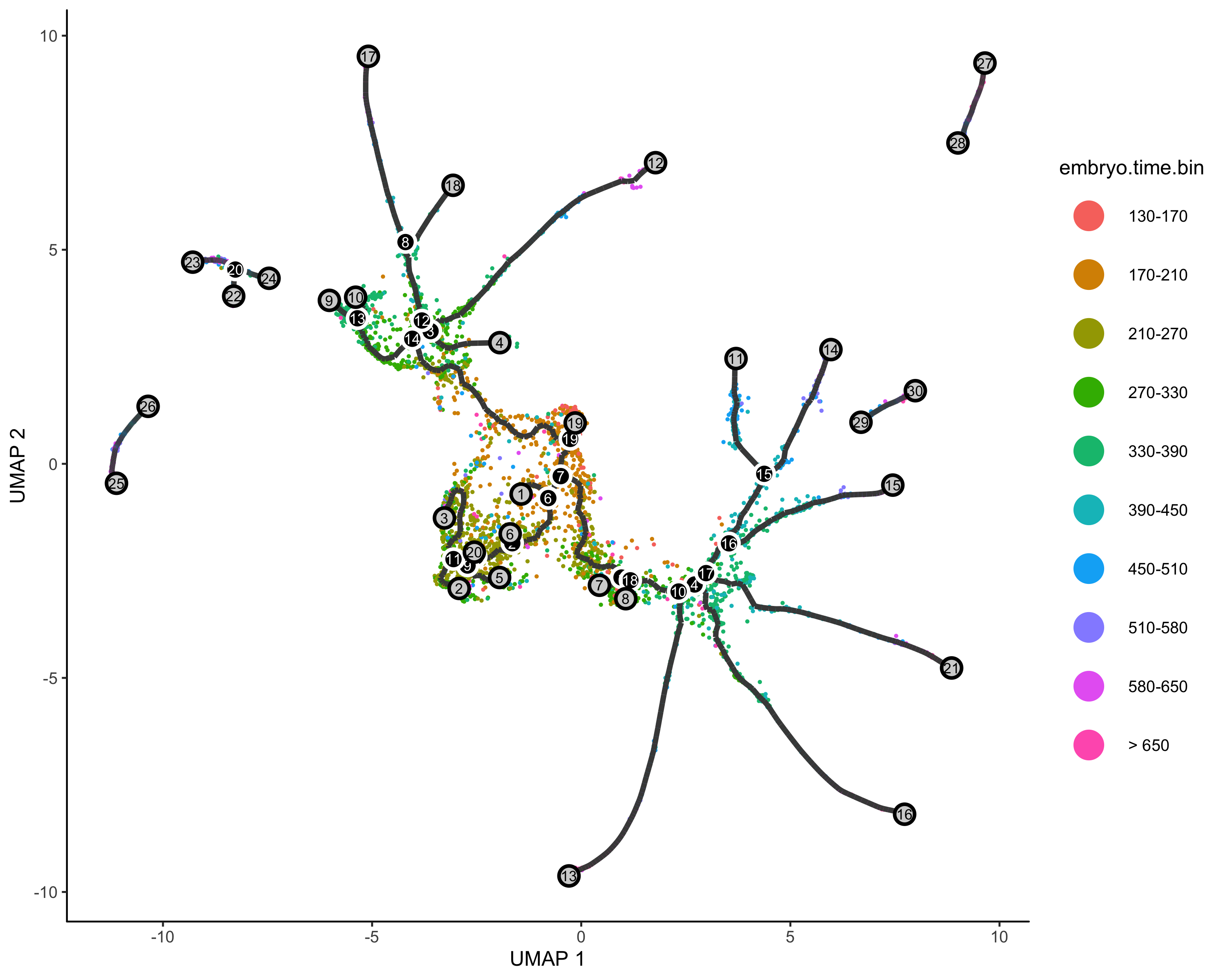
定义起始节点&&伪时间分析
起始节点可以基于现有的知识,比如你已经将细胞类型注释好了,其中某种细胞就是起始的早期细胞,我们就可以把它作为root,如果不知道,我们只能使用1在的细胞群作为起始了
一个有用的定义根节点的函数
1 | get_earliest_principal_node <- function(cds, time_bin="Dendritic.cells.activated"){ |
定义根节点
1 | #order cell |
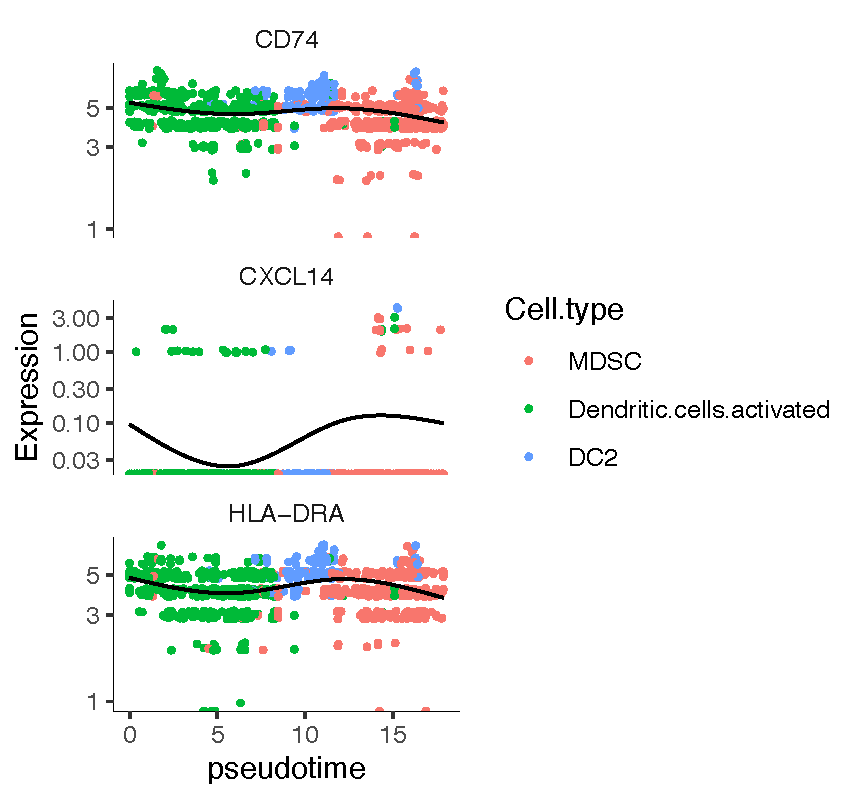
在基因层面探索伪时间
寻找随时间变异的基因
1 | diff_gene <- graph_test(cds, neighbor_graph="principal_graph", cores=4) |
在cds对象中寻找基因并作图,为了看出变化,可以选择case组中的表达量
1 | CASE_genes <- c("CD74", "CXCL14", "HLA-DRA") |
教程到此结束,原创禁止转载
文中图片大部分取自官网,仅作为示例。









