文章已经过时,请去官网查阅相关文档
本教程介绍了Kang等人(2017)的两组PBMC的对齐方式。在该实验中,将PBMC分为刺激组和对照组,并用干扰素β治疗刺激组。对干扰素的反应引起细胞类型特异性基因表达的变化,这使得对所有数据进行联合分析变得困难,并且细胞按刺激条件和细胞类型聚类。在这里,我们证明了我们的整合策略,如Stuart和Butler等人(2018年)所述,用于执行整合分析以促进常见细胞类型的鉴定并进行比较分析。尽管此示例演示了两个数据集(条件)的集成,但这些方法已扩展到多个数据集。这个工作流程提供了整合四个胰岛数据集的示例。
整合目标
以下教程旨在概述使用Seurat集成过程可能进行的复杂细胞类型的比较分析。在这里,我们解决了三个主要目标:
- 识别两个数据集中都存在的单元格类型
- 获得在对照和刺激细胞中均保守的细胞类型标记
- 比较数据集以找到对刺激的细胞类型特异性反应
设置Seurat对象
为方便起见,我们通过SeuratData软件包分发此数据集。
library(Seurat) |
执行整合
然后,我们使用FindIntegrationAnchors函数来识别锚点,该函数将Seurat对象的列表作为输入,并使用这些锚点将两个数据集与集成在一起。
immune.anchors <- FindIntegrationAnchors(object.list = ifnb.list, dims = 1:20) |
进行综合分析
现在,我们可以在所有单元上运行单个集成分析!
DefaultAssay(immune.combined) <- "integrated" |
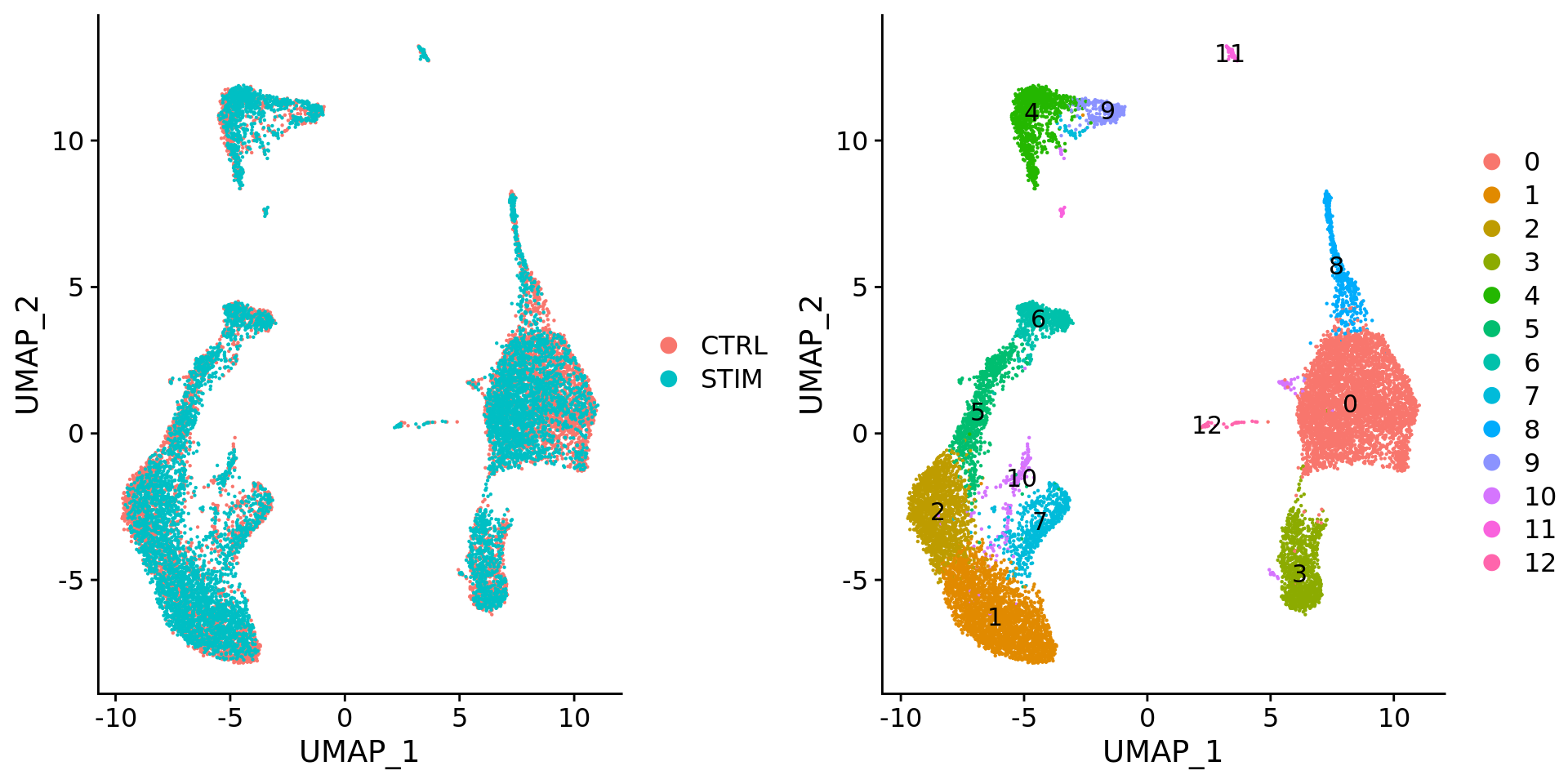
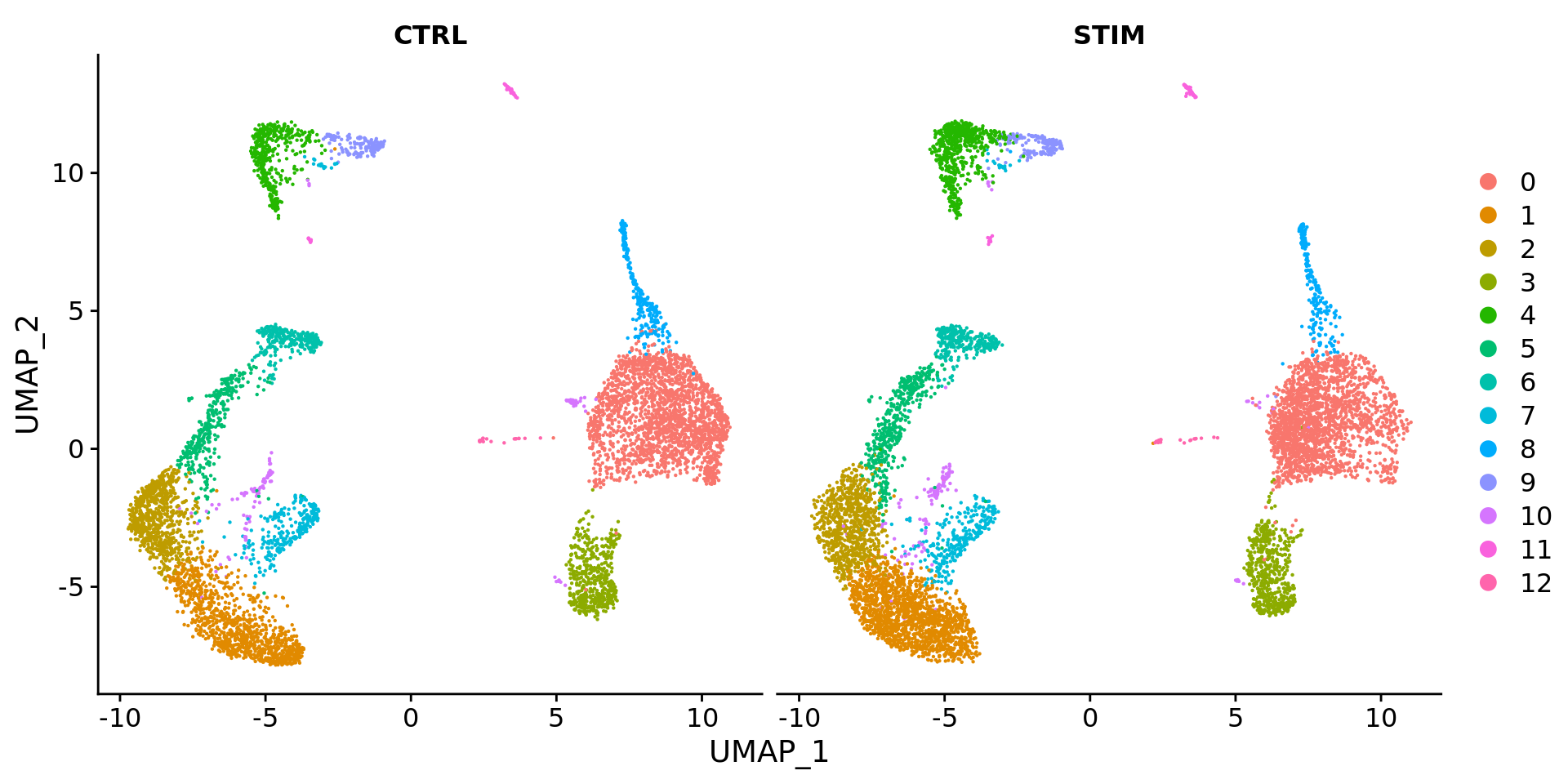
为了并排可视化这两个条件,我们可以使用split.by参数来显示每个以聚类着色的条件。
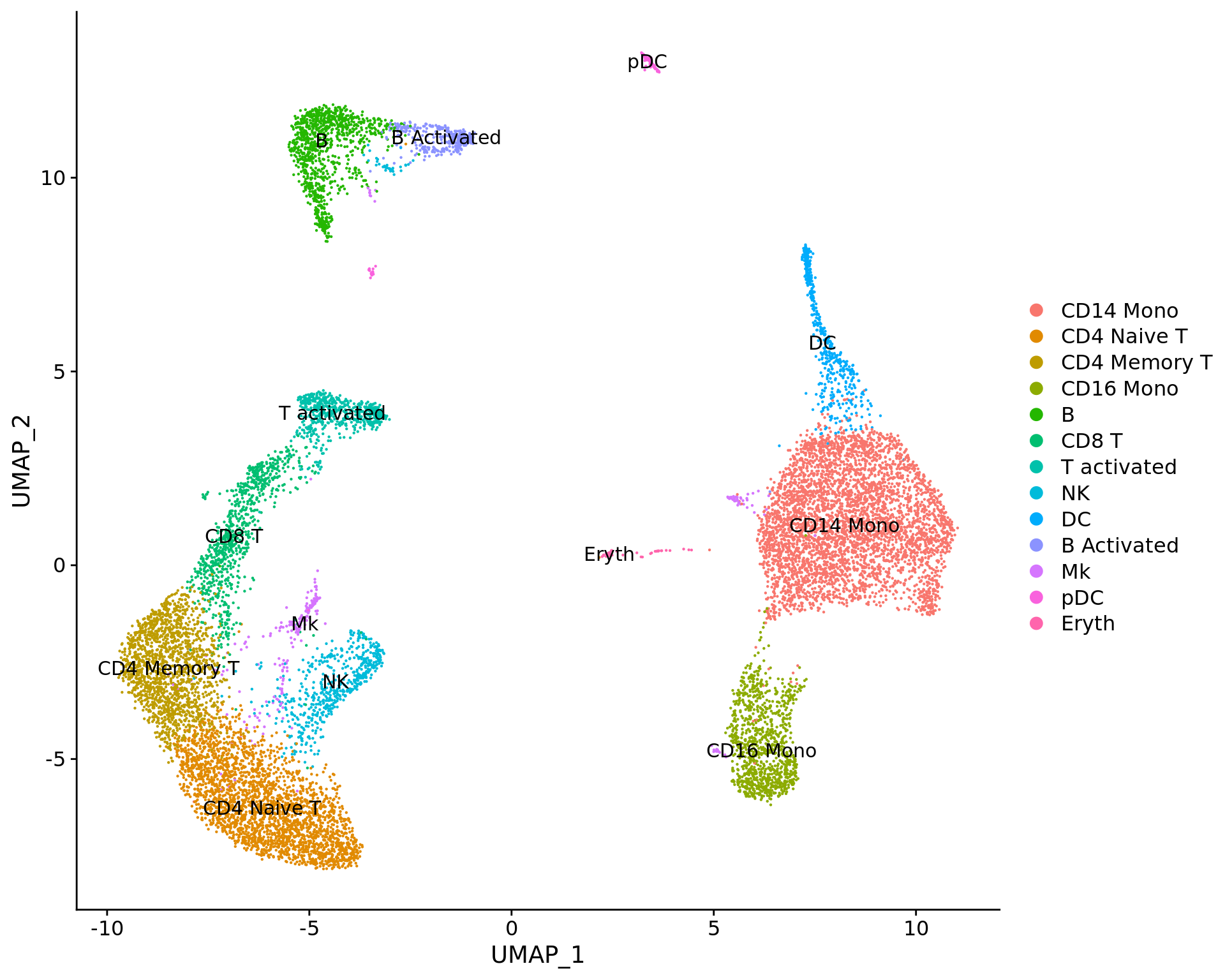
DimPlot(immune.combined, reduction = "umap", split.by = "stim") |
识别保守的细胞类型标记
为了确定跨条件保守的规范细胞类型标记基因,我们提供了该FindConservedMarkers功能。此功能对每个数据集/组执行差异基因表达测试,并使用MetaDER软件包中的荟萃分析方法组合p值。例如,无论簇7中的刺激条件如何,我们都可以计算出保守标记的基因(NK细胞)。
DefaultAssay(immune.combined) <- "RNA" |
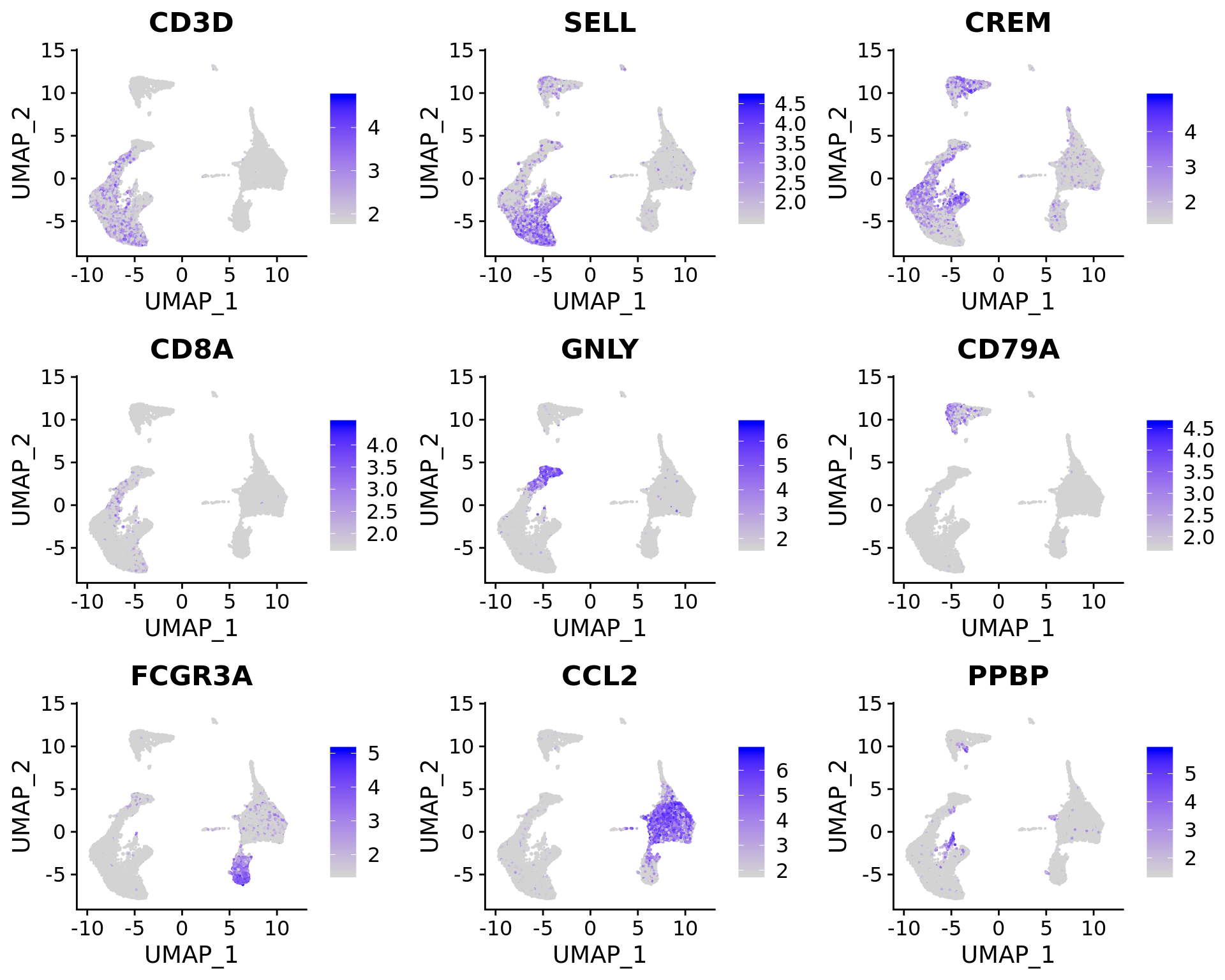
我们可以为每个簇探索这些标记基因,并使用它们将我们的簇注释为特定的细胞类型。
FeaturePlot(immune.combined, features = c("CD3D", "SELL", "CREM", "CD8A", "GNLY", "CD79A", "FCGR3A", |
immune.combined <- RenameIdents(immune.combined, `0` = "CD14 Mono", `1` = "CD4 Naive T", `2` = "CD4 Memory T", |
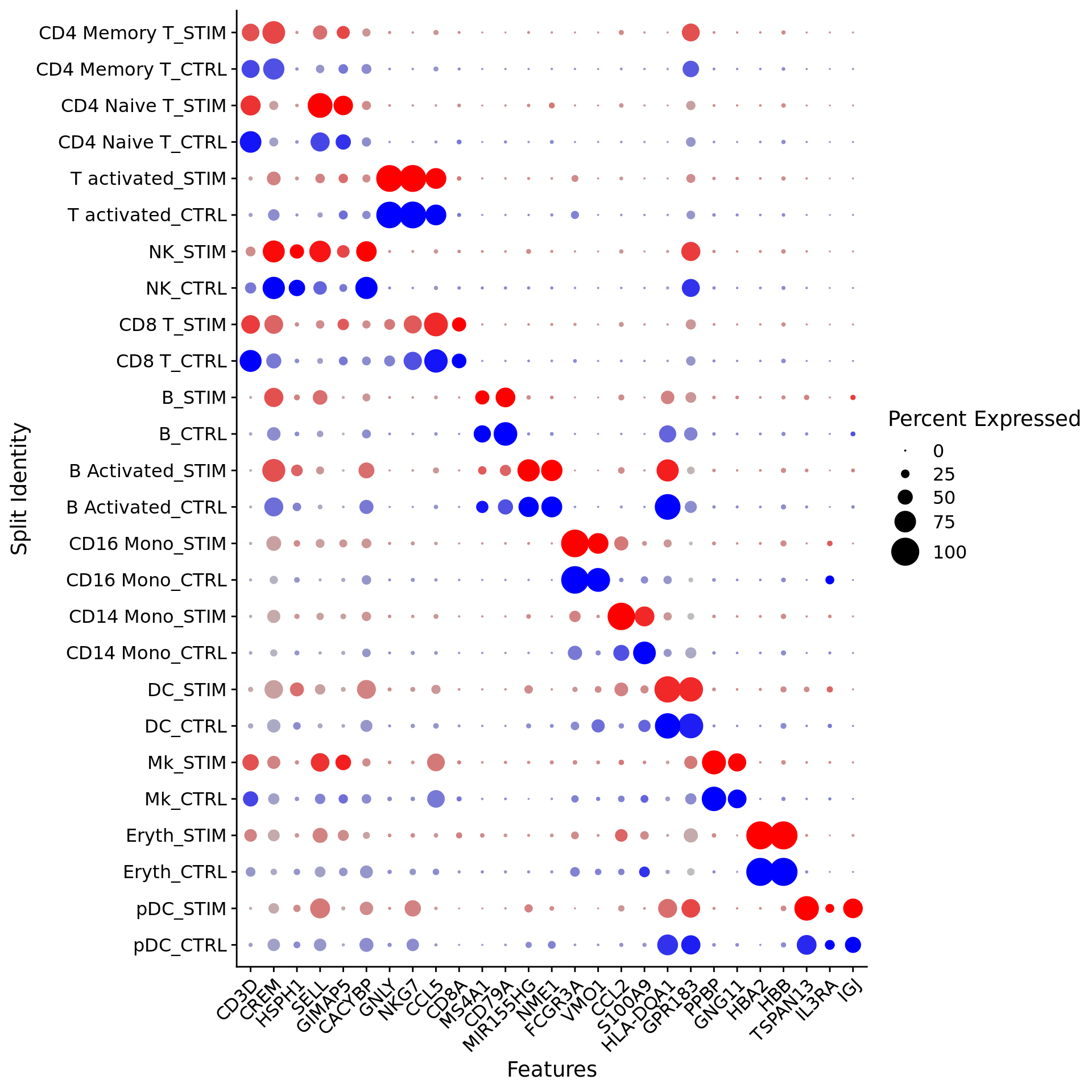
DotPlot带有split.by参数的函数可用于查看各种条件下保守的细胞类型标记,显示表达水平和表达任何给定基因的簇中细胞的百分比。在这里,我们为13个簇中的每个簇绘制2-3个强标记基因。
Idents(immune.combined) <- factor(Idents(immune.combined), levels = c("Mono/Mk Doublets", "pDC", |
跨条件鉴定差异表达基因
现在我们已经排列了刺激细胞和对照细胞,我们可以开始进行比较分析,并观察刺激引起的差异。广泛观察这些变化的一种方法是绘制受刺激细胞和对照细胞的平均表达,并在散点图上寻找视觉异常值的基因。在这里,我们采用受刺激的和对照的原始T细胞和CD14单核细胞群体的平均表达,并生成散点图,突出显示对干扰素刺激表现出戏剧性反应的基因。
library(ggplot2) |
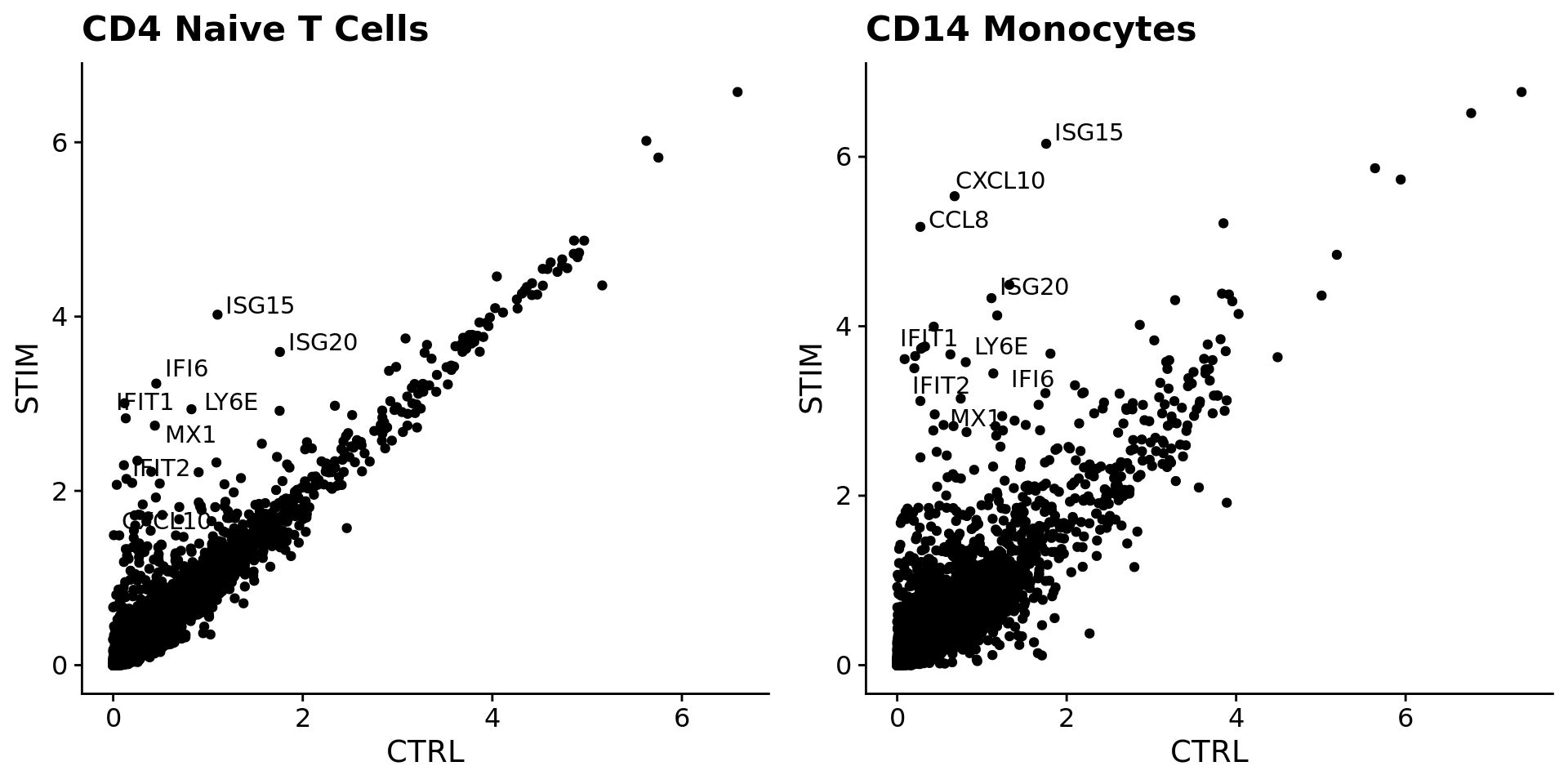
如您所见,许多相同的基因在这两种细胞类型中均被上调,可能代表保守的干扰素应答途径。
因为我们有信心在各种情况下都能识别出常见的细胞类型,所以我们可以询问相同类型的细胞在不同条件下会改变哪些基因。首先,我们在meta.data插槽中创建一列,以保存细胞类型和刺激信息,并将当前标识切换到该列。然后,我们使用它FindMarkers来找到受刺激的B细胞和对照B细胞之间不同的基因。请注意,此处显示的许多顶级基因与我们之前绘制的核心干扰素应答基因相同。此外,我们看到的诸如CXCL10的基因对单核细胞和B细胞干扰素的反应也具有特异性,在该列表中也显示出很高的意义。
immune.combined$celltype.stim <- paste(Idents(immune.combined), immune.combined$stim, sep = "_") |
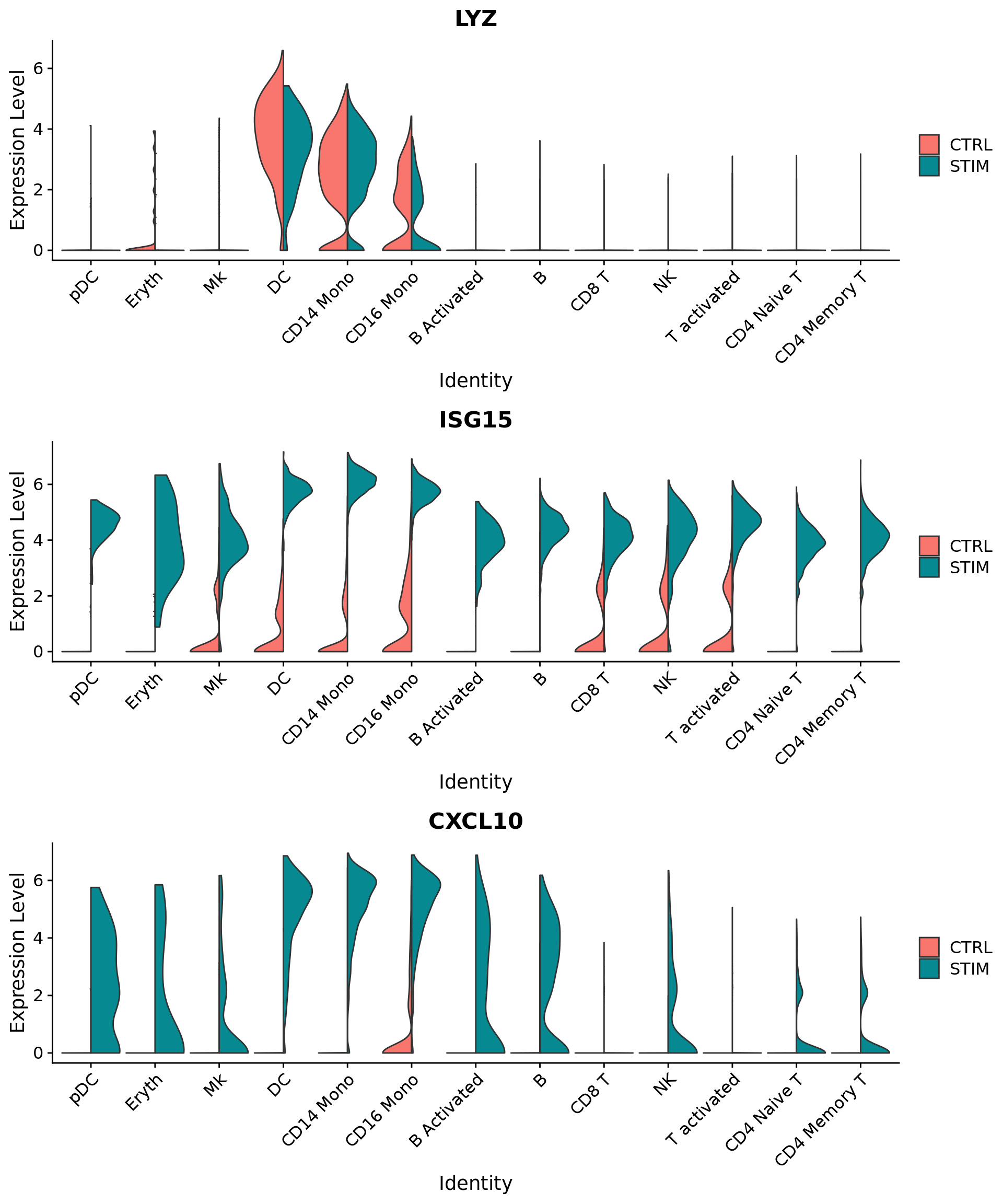
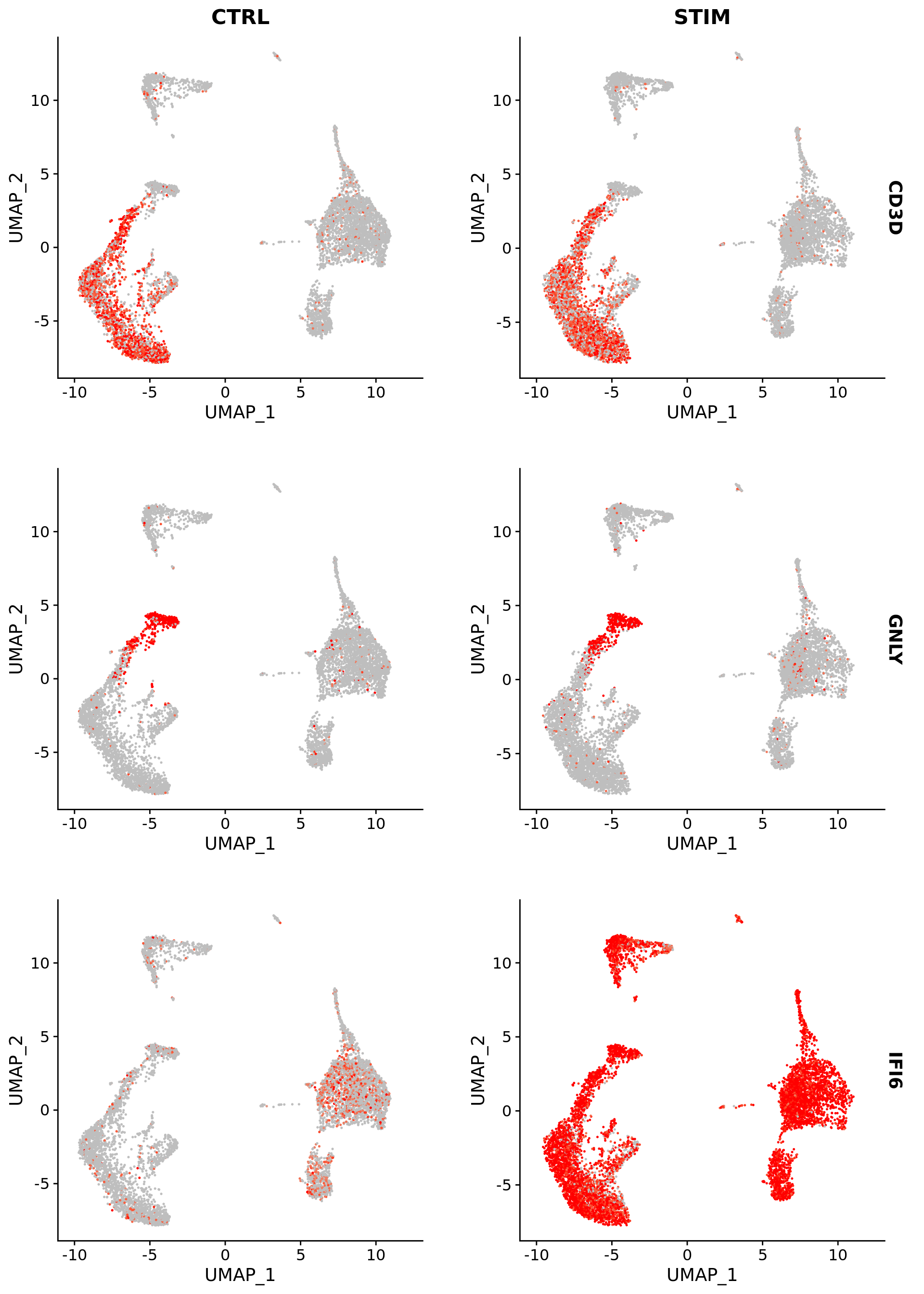
可视化基因表达中这些变化的另一种有用方法是split.by选择FeaturePlot或VlnPlot功能。这将显示给定基因列表的FeaturePlots,并按分组变量(此处为刺激条件)进行划分。诸如CD3D和GNLY之类的基因是典型的细胞类型标记(对于T细胞和NK / CD8 T细胞),实际上不受干扰素刺激的影响,并且在对照组和受刺激组中显示出相似的基因表达模式。另一方面,IFI6和ISG15是核心干扰素应答基因,因此在所有细胞类型中均被上调。最后,CD14和CXCL10是显示细胞类型特异性干扰素应答的基因。CD14单核细胞受刺激后,CD14表达下降,这可能导致在有监督的分析框架中进行错误分类,从而强调了整合分析的价值。
FeaturePlot(immune.combined, features = c("CD3D", "GNLY", "IFI6"), split.by = "stim", max.cutoff = 3, |
plots <- VlnPlot(immune.combined, features = c("LYZ", "ISG15", "CXCL10"), split.by = "stim", group.by = "celltype", |